認知課題の解析データセット作成
1 統計解析よりも解析データセット作成が大切!
解析の再生可能性を高めるうえで,統計解析自体よりも解析データセット作成が大切です。特に認知課題から得られるデータは,統計解析にかけるまでのデータセットの作成段階で色々と操作が必要です(不要なものを除外したり,複数の参加者のデータを統合したり,変数に処理を加えたりなど)。その際に,ミスが生じがちです。これまで学生指導していて,データ解析でミスが混入する危険性が高いのがここです。この段階でのミスは発覚しにくく,後で追えないこともあり,最もデータ処理上気をつけなくてはいけない部分です。以下では,これらの問題をクリアするために,Rを用いた解析データセットの作成方法について解説します。
1.1 解析データセット作成ではExcelを使わない
最初に,解析データセットの作成では,Excelの使用を禁止します。ExcelもSPSSもマウスを使って操作をしますが,そのような操作はミスが生じやすく,その操作過程の記録が残らないことも多いので,注意が必要です(操作過程が残っていても追いにくい面もあります)。例えば以下のようなことがあります。
- 例1) 各参加者のデータを解析用エクセルファイルにコピー&ペーストしていたが,参加者Aのデータをコピー&ペーストしてから,参加者Bのデータをコピー&ペーストしたが,実際にはコピーができておらず,参加者Aのデータが2回入ってしまった。
こういうことがあると,解析で得られた結果は真実とは違ったものになります。100%ミスせずにExcelでコピー&ペーストできる保証はありません。
- 例2) Excel上でデータの確認のために,特定の変数の値で並び替えたが,その並び替えが特定の変数だけに指定されており,参加者の本当の値とは違う値が入ることになった。
こういうことがあると,解析で得られた結果は真実とは違ったものになります。ソートに限らず,なんらかの処理をエクセル上でしてしまうと,それが本当にただしい処理をしたのか後で確認できなくなることがあります。
1.2 解析データセット作成ではRを使おう
認知課題解析用データセットの作成は,以下の原則を守りつつ行う必要があります。
- オリジナルの生データを直接操作しない(必ず別途バックアップとっておく)
- オリジナルの生データから必要な情報をマウス操作ではない形で抽出する
- オリジナルの生データを1つのファイルにまとめて,解析可能なフォーマットに変更する
- 上記の操作について作業ログが残り,何度でもやり直せる
このような条件を満たすもので,心理学科の学生にも使いやすいものとしてはRがあります。解析データセット作成では,Rを使いましょう!以下では,(1)jsPsychを用いたWEB実験で得られたJSONファイルの処理(JATOSとPavloviaで分けています),(2)PsychoPyを用いた実験で得られたcsvファイルの処理について解説します。なお,RやRstudioの基本的な使い方については,R言語の基本と解析のパッケージ化で学んでいることを前提とします。
2 jsPsychを用いたWEB実験で得られたJSONファイルの処理(JATOS編)
2.1 データとフォルダの準備
jsPsychで作成しJATOSで収集したWEB実験のサンプルデータをクリックして,ダウンロードして,自分のパソコンの好きな場所に解凍・展開してください。解凍・展開すると,jatos_results_20210513054636.txtというファイルが出てくると思います(国里が 逆転学習課題に2回取り組んだデータです)。RstudioのWorking Directoryにこのjatos_results_20210513054636.txtを置くか,jatos_results_20210513054636.txtのある場所をRstudioのWorking Directoryにしてください。
2.2 使用するRパッケージ
- JSONファイルの読み込みのためにjsonlite,データの整形のためにtidyverseを使用します。
library(jsonlite)
library(tidyverse)2.3 JSONファイルの読み込み
JATOSで収集してエクスポートしたデータはJSON形式ですが,そのままでは,jsonliteパッケージを使って読み込めません。そこで,lab.jsで解説されている方法を使います。以下のように,read_file()で読み込んだ内容を,str_split()を使って改行で区切ります(これによって参加者ごとのデータで区切られる)。するとリスト化されちゃうので,first()を使ってデータだけ取り出します。完全ではないデータの場合,”空白”になるので,それを,discard() で除外します。最後に,map_dfr(fromJSON, flatten=T)で各参加者のJSON形式のデータを読み込んでRで使えるようにします。
data_raw <- read_file('jatos_results_20210513054636.txt') %>%
str_split('\n') %>%
first() %>%
discard(function(x) x == '') %>%
map_dfr(fromJSON, flatten=T)2.4 読み込んだJSONファイルの整形
JSONから読み込んだだけだと解析では使えないので,以下でデータ整形をしてきます。as_tibble()でtibble化します。今回の実験の場合,html-button-response以外は教示やフィードバックなので,filter(trial_type == “html-button-response”)でhtml-button-responseにしぼります。次に,参加者が受けた報酬を変数にするために,mutate(reward = ifelse(response == 0,reward_s1,reward_s2))でresponseで0の場合は(S1を選んだ場合),reward_s1,それ以外はreward_s2をrewardにいれます。次のgroup_by(id) %>% mutate(trial = row_number()) %>% ungroup() %>% は,各参加者のidごとに試行番号(trial)をつけるために,idでグループ化して,row_number()をつかってtrial番号をつくって,最後にグループ化をといています。mutate(response = as.numeric(response),reward = as.numeric(reward))は,一部が文字型とかになっているので,数値型にしています。最後に,その後の解析必要なものをselect(id,trial,response,reward,rt) で選んでいます。
data <- data_raw %>%
as_tibble(validate = F) %>%
filter(trial_type == "html-button-response") %>%
mutate(reward = ifelse(response == 0,reward_s1,reward_s2)) %>%
group_by(id) %>%
mutate(trial = row_number()) %>%
ungroup() %>%
mutate(response = as.numeric(response),reward = as.numeric(reward)) %>%
select(id,trial,response,reward,rt) 2.5 データのチェック
上記で整形したデータをプロットしてみましょう。各trialのresponseをプロットしてみます。逆転学習課題は,最初はS1のほうが報酬がよく出て,後半はS2のほうが報酬がよく出ました。それに合わせて,反応(response)も前半は0(S1),後半は1(S2)が多くなっています。
data %>%
filter(id == "4dk59k") %>%
filter(!is.na(response)) %>%
ggplot(aes(x = trial, y = response)) +
geom_point()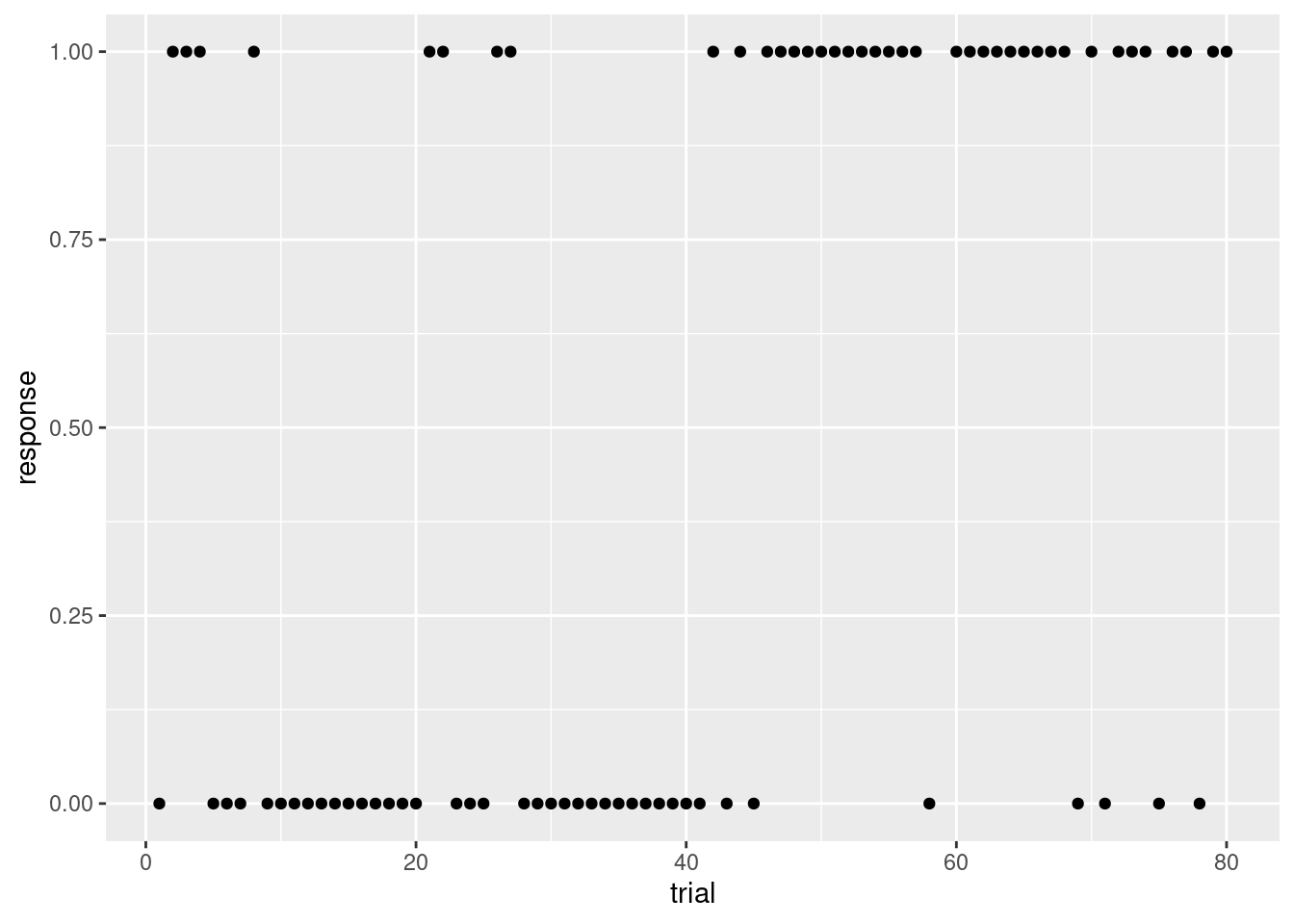
2.6演習課題1
ゼミ生で協力して,jsPsychで逆転学習課題を作成して,JATOSにホスティングして,10名ほどデータを集めてみましょう。その結果をRで読み込んで処理をして,可視化してみましょう。
3 jsPsychを用いたWEB実験で得られたJSONファイルの処理(Pavlovia編)
3.1 データとフォルダの準備
jsPsychで作成しPavloviaで収集したWEB実験のサンプルデータをクリックして,ダウンロードして,自分のパソコンの好きな場所に解凍・展開してください。解凍・展開すると,stroop.jsonというファイルが出てくると思います(国里がstroop課題に3回取り組んだデータです)。RstudioのWorking Directoryにこのstroop.jsonを置くか,stroop.jsonのある場所をRstudioのWorking Directoryにしてください。
3.2 使用するRパッケージ
- JSONファイルの読み込みのためにjsonlite,データの整形のためにtidyverseを使用します。
library(jsonlite)
library(tidyverse)3.3 JSONファイルの読み込み
ダウンロードしたJSONファイルを読み込む際には,以下のように,jsonliteパッケージのfromJSON()関数を使います。データ名は,list形式のdatabaseデータなので,db_listとしました。
db_list <- fromJSON("stroop.json")- glimpse()で,読み込んだJSONファイルの構造をチェックします。このデータは,私が3回Stroop課題に取り組んだものです。listは参加者で構成されていますので,3つあります。そして,それぞれの下に実験データのdata.frameがあります。
glimpse(db_list)## List of 3
## $ 2019_8_22_23_48_59_fxlmh9SL:List of 1
## ..$ data:'data.frame': 19 obs. of 10 variables:
## .. ..$ internal_node_id: chr [1:19] "0.0-0.0" "0.0-1.0" "0.0-2.0-0.0" "0.0-2.0-0.1" ...
## .. ..$ key_press : int [1:19] 13 68 68 68 75 68 74 74 75 70 ...
## .. ..$ rt : num [1:19] 390003 7183 706 816 1119 ...
## .. ..$ stimulus : chr [1:19] "<p><span style='font-size:20pt;'>実験にお越しいただき,ありがとうございます!!!</span></p><p>キーボードのキーを"| __truncated__ "<p style='text-align:left'>この課題では, 以下のような色のついた単語を見ていただきます。</p><p style='color:red;"| __truncated__ "<p style='color:red;font-size:60pt;'>赤</p>" "<p style='color:red;font-size:60pt;'>赤</p>" ...
## .. ..$ time_elapsed : int [1:19] 390012 397196 398906 401142 404260 406947 408927 411447 412857 414985 ...
## .. ..$ trial_index : int [1:19] 0 1 2 3 4 5 6 7 8 9 ...
## .. ..$ trial_type : chr [1:19] "html-keyboard-response" "html-keyboard-response" "html-keyboard-response" "html-keyboard-response" ...
## .. ..$ correct : logi [1:19] NA NA TRUE TRUE TRUE TRUE ...
## .. ..$ response : chr [1:19] NA NA "d" "d" ...
## .. ..$ stim_type : chr [1:19] NA NA "congruent" "congruent" ...
## $ 2019_8_22_23_56_32_juStKRbZ:List of 1
## ..$ data:'data.frame': 19 obs. of 10 variables:
## .. ..$ internal_node_id: chr [1:19] "0.0-0.0" "0.0-1.0" "0.0-2.0-0.0" "0.0-2.0-0.1" ...
## .. ..$ key_press : int [1:19] 13 68 68 70 74 70 75 75 68 68 ...
## .. ..$ rt : num [1:19] 2694 2233 554 962 1573 ...
## .. ..$ stimulus : chr [1:19] "<p><span style='font-size:20pt;'>実験にお越しいただき,ありがとうございます!!!</span></p><p>キーボードのキーを"| __truncated__ "<p style='text-align:left'>この課題では, 以下のような色のついた単語を見ていただきます。</p><p style='color:red;"| __truncated__ "<p style='color:red;font-size:60pt;'>赤</p>" "<p style='color:blue;font-size:60pt;'>赤</p>" ...
## .. ..$ time_elapsed : int [1:19] 2696 4930 6491 8290 10571 12205 14231 15730 18340 19780 ...
## .. ..$ trial_index : int [1:19] 0 1 2 3 4 5 6 7 8 9 ...
## .. ..$ trial_type : chr [1:19] "html-keyboard-response" "html-keyboard-response" "html-keyboard-response" "html-keyboard-response" ...
## .. ..$ correct : logi [1:19] NA NA TRUE TRUE TRUE TRUE ...
## .. ..$ response : chr [1:19] NA NA "d" "f" ...
## .. ..$ stim_type : chr [1:19] NA NA "congruent" "incongruent" ...
## $ 2019_8_22_23_57_16_8EygxO3q:List of 1
## ..$ data:'data.frame': 19 obs. of 10 variables:
## .. ..$ internal_node_id: chr [1:19] "0.0-0.0" "0.0-1.0" "0.0-2.0-0.0" "0.0-2.0-0.1" ...
## .. ..$ key_press : int [1:19] 13 68 74 74 70 NA 68 68 70 75 ...
## .. ..$ rt : num [1:19] 1413 1903 1848 603 636 ...
## .. ..$ stimulus : chr [1:19] "<p><span style='font-size:20pt;'>実験にお越しいただき,ありがとうございます!!!</span></p><p>キーボードのキーを"| __truncated__ "<p style='text-align:left'>この課題では, 以下のような色のついた単語を見ていただきます。</p><p style='color:red;"| __truncated__ "<p style='color:green;font-size:60pt;'>赤</p>" "<p style='color:green;font-size:60pt;'>赤</p>" ...
## .. ..$ time_elapsed : int [1:19] 1414 3319 6169 8193 10684 14600 17180 18381 21319 23915 ...
## .. ..$ trial_index : int [1:19] 0 1 2 3 4 5 6 7 8 9 ...
## .. ..$ trial_type : chr [1:19] "html-keyboard-response" "html-keyboard-response" "html-keyboard-response" "html-keyboard-response" ...
## .. ..$ correct : logi [1:19] NA NA TRUE TRUE TRUE FALSE ...
## .. ..$ response : chr [1:19] NA NA "j" "j" ...
## .. ..$ stim_type : chr [1:19] NA NA "incongruent" "incongruent" ...3.4 読み込んだJSONファイルの整形
- 読み込んだJSONデータをtidyverseのas.tibble()関数を使ってデータフレームにします(データフレームなので,db_dfという名前にしました)。すると,3つの参加者が変数になって,変数の下にdata.frameができました。
db_df <- as_tibble(db_list, validate = F)
db_df## # A tibble: 1 × 3
## `2019_8_22_23_48_59_fxlmh9SL` `2019_8_22_23_56_32_juStKRbZ` `2019_8_22_23_57…`
## <named list> <named list> <named list>
## 1 <df [19 × 10]> <df [19 × 10]> <df [19 × 10]>- Rでは,第1行目に変数名があり,各変数名の下にデータがある形のものを扱います。上記の形式は,参加者名が列ではなく行になってしまっているので(wide型になっている),困ります。そこで,tidyverseのgather()関数を使って,wide型とlong型にします。gather()関数では,gather(データ,wide型で変数名になっているものを格納するlong型での変数名,wide型でデータになっているものをlong型のデータにしたときの変数名)という感じで引数を設定します。なお,db_dfに代入すると上書きされるので,db_df2に代入をします。IDの列とdataの列ができていい感じです。
db_df2 <- gather(db_df, ID, data)
db_df2## # A tibble: 3 × 2
## ID data
## <chr> <list>
## 1 2019_8_22_23_48_59_fxlmh9SL <df [19 × 10]>
## 2 2019_8_22_23_56_32_juStKRbZ <df [19 × 10]>
## 3 2019_8_22_23_57_16_8EygxO3q <df [19 × 10]>- gatherでいい感じになってきましたが,各参加者のdataは,値ではなくてdata.frameがネストしちゃっているので,unnestします。これで,とりあえず解析データセットが完成しました。
db_df3 <-unnest(db_df2,cols = data)
db_df3## # A tibble: 57 × 11
## ID internal_node_id key_press rt stimulus time_elapsed trial_index
## <chr> <chr> <int> <dbl> <chr> <int> <int>
## 1 2019_8_2… 0.0-0.0 13 3.90e5 <p><spa… 390012 0
## 2 2019_8_2… 0.0-1.0 68 7.18e3 <p styl… 397196 1
## 3 2019_8_2… 0.0-2.0-0.0 68 7.06e2 <p styl… 398906 2
## 4 2019_8_2… 0.0-2.0-0.1 68 8.16e2 <p styl… 401142 3
## 5 2019_8_2… 0.0-2.0-0.2 75 1.12e3 <p styl… 404260 4
## 6 2019_8_2… 0.0-2.0-0.3 68 8.34e2 <p styl… 406947 5
## 7 2019_8_2… 0.0-2.0-0.4 74 1.06e3 <p styl… 408927 6
## 8 2019_8_2… 0.0-2.0-0.5 74 1.15e3 <p styl… 411447 7
## 9 2019_8_2… 0.0-2.0-0.6 75 7.51e2 <p styl… 412857 8
## 10 2019_8_2… 0.0-2.0-0.7 70 8.04e2 <p styl… 414985 9
## # … with 47 more rows, and 4 more variables: trial_type <chr>, correct <lgl>,
## # response <chr>, stim_type <chr>- これでOKな気もしますが,不要な変数がいくつかありますし,教示などの参加者の反応が入ってないデータも不要だったりしますので,整形作業が必要です。まずは,その後の解析で使うであろうID,trial_index,stim_type,response,correct,rtのみを,tidyverseのselect()関数を使って選択します。なお,ここで”%>% “というなぞのコマンドがでてきましたが,これはパイプ演算子です(tidyverseパッケージを読み込むと使えます)。”A %>% B”は,Aを関数Bの第1引数(多くの関数の場合,データ)にいれるという意味です。大した機能ではなさそうですが,処理を重ねる場合に,関数から関数にデータを引き継いで処理が書けるので,わかりやすくかけるというメリットがあります(最後にその例を示します)。
db_df4 <-db_df3 %>%
select(ID,trial_index,stim_type,response,correct,rt)
db_df4## # A tibble: 57 × 6
## ID trial_index stim_type response correct rt
## <chr> <int> <chr> <chr> <lgl> <dbl>
## 1 2019_8_22_23_48_59_fxlmh9SL 0 <NA> <NA> NA 390003.
## 2 2019_8_22_23_48_59_fxlmh9SL 1 <NA> <NA> NA 7183.
## 3 2019_8_22_23_48_59_fxlmh9SL 2 congruent d TRUE 706.
## 4 2019_8_22_23_48_59_fxlmh9SL 3 congruent d TRUE 816.
## 5 2019_8_22_23_48_59_fxlmh9SL 4 incongruent k TRUE 1119.
## 6 2019_8_22_23_48_59_fxlmh9SL 5 congruent d TRUE 834.
## 7 2019_8_22_23_48_59_fxlmh9SL 6 incongruent j TRUE 1056.
## 8 2019_8_22_23_48_59_fxlmh9SL 7 incongruent j TRUE 1151.
## 9 2019_8_22_23_48_59_fxlmh9SL 8 incongruent k TRUE 751.
## 10 2019_8_22_23_48_59_fxlmh9SL 9 incongruent f TRUE 804.
## # … with 47 more rows- trial_indexの0はウェルカムメッセージ,1は教示,18は課題終了の教示です。これらは不要ですので,tidyverseのfilter()関数を使って除外します(filter関数は特定の条件のデータを残す関数ですが,!=と指定すれば,該当するデータを除外してくれます)。
db_df5 <-db_df4 %>%
filter(trial_index !=0) %>% #ウェルカムメッセージは不要
filter(trial_index !=1) %>% #教示は不要
filter(trial_index !=18) #最後の教示も不要
db_df5## # A tibble: 48 × 6
## ID trial_index stim_type response correct rt
## <chr> <int> <chr> <chr> <lgl> <dbl>
## 1 2019_8_22_23_48_59_fxlmh9SL 2 congruent d TRUE 706.
## 2 2019_8_22_23_48_59_fxlmh9SL 3 congruent d TRUE 816.
## 3 2019_8_22_23_48_59_fxlmh9SL 4 incongruent k TRUE 1119.
## 4 2019_8_22_23_48_59_fxlmh9SL 5 congruent d TRUE 834.
## 5 2019_8_22_23_48_59_fxlmh9SL 6 incongruent j TRUE 1056.
## 6 2019_8_22_23_48_59_fxlmh9SL 7 incongruent j TRUE 1151.
## 7 2019_8_22_23_48_59_fxlmh9SL 8 incongruent k TRUE 751.
## 8 2019_8_22_23_48_59_fxlmh9SL 9 incongruent f TRUE 804.
## 9 2019_8_22_23_48_59_fxlmh9SL 10 congruent d TRUE 768.
## 10 2019_8_22_23_48_59_fxlmh9SL 11 incongruent j TRUE 1206.
## # … with 38 more rows- よく見たら,正誤の変数のcorrectは,TRUE・FALSEになっていますね。そこで,tidyverseのmutate()関数を使って,TRUE・FALSE型を数値型にした変数を別途作ります(mutate()関数は変数を作成するときに使えます)。
db_df6 <-db_df5 %>%
mutate(correct2 = as.numeric(correct))
db_df6## # A tibble: 48 × 7
## ID trial_index stim_type response correct rt correct2
## <chr> <int> <chr> <chr> <lgl> <dbl> <dbl>
## 1 2019_8_22_23_48_59_fxl… 2 congruent d TRUE 706. 1
## 2 2019_8_22_23_48_59_fxl… 3 congruent d TRUE 816. 1
## 3 2019_8_22_23_48_59_fxl… 4 incongru… k TRUE 1119. 1
## 4 2019_8_22_23_48_59_fxl… 5 congruent d TRUE 834. 1
## 5 2019_8_22_23_48_59_fxl… 6 incongru… j TRUE 1056. 1
## 6 2019_8_22_23_48_59_fxl… 7 incongru… j TRUE 1151. 1
## 7 2019_8_22_23_48_59_fxl… 8 incongru… k TRUE 751. 1
## 8 2019_8_22_23_48_59_fxl… 9 incongru… f TRUE 804. 1
## 9 2019_8_22_23_48_59_fxl… 10 congruent d TRUE 768. 1
## 10 2019_8_22_23_48_59_fxl… 11 incongru… j TRUE 1206. 1
## # … with 38 more rows- 上書きしなようにすると,db_dfが6まで増えてしまいました。ここで,“%>%”が役に立ちます。関数を何度も使って処理を重ねていく場合に,以下のように”%>%“でつないでいくと,一気にまとめて処理できますし,見た目も何をしているかわかりやすいです。なにか処理をした場合に,#を使ってコメントを残しておくとわかりやすいです。なお,mutate関数の位置を少し変えて,select関数適用後にlogical型のcorrectが残らないように変更しています。
db_df <- db_list %>%
as_tibble(validate = F) %>% #データフレーム化
gather(ID, data) %>% #ロング化
unnest(cols = data) %>% #ネストやめる
mutate(correct2 = as.numeric(correct)) %>% #正誤反応はlogical型からnumeric型に
select(ID,trial_index,stim_type,response,correct2,rt) %>% #使う変数選択
filter(trial_index !=0) %>% #ウェルカムメッセージを除外
filter(trial_index !=1) %>% #教示を除外
filter(trial_index !=18) #終了時の教示を除外
db_df## # A tibble: 48 × 6
## ID trial_index stim_type response correct2 rt
## <chr> <int> <chr> <chr> <dbl> <dbl>
## 1 2019_8_22_23_48_59_fxlmh9SL 2 congruent d 1 706.
## 2 2019_8_22_23_48_59_fxlmh9SL 3 congruent d 1 816.
## 3 2019_8_22_23_48_59_fxlmh9SL 4 incongruent k 1 1119.
## 4 2019_8_22_23_48_59_fxlmh9SL 5 congruent d 1 834.
## 5 2019_8_22_23_48_59_fxlmh9SL 6 incongruent j 1 1056.
## 6 2019_8_22_23_48_59_fxlmh9SL 7 incongruent j 1 1151.
## 7 2019_8_22_23_48_59_fxlmh9SL 8 incongruent k 1 751.
## 8 2019_8_22_23_48_59_fxlmh9SL 9 incongruent f 1 804.
## 9 2019_8_22_23_48_59_fxlmh9SL 10 congruent d 1 768.
## 10 2019_8_22_23_48_59_fxlmh9SL 11 incongruent j 1 1206.
## # … with 38 more rows3.5 データのチェック
上記の作業で解析データセットができました。簡単に3名の参加者の条件ごと(ストループ課題での色と文字の一致条件と不一致条件)の正答率と平均反応時間を見てみましょう。tidyverseを使えば,group_byとsummariseで簡単にこういう整理ができます。なお,参加者のIDがちょっと長すぎたので,mutateを使ってIDをtime1,time2,time3に書き換えたidという変数を作って,使っています。このデータは,私が適当に3回回答したものですので,time1~3というidにしました。結果をみると,全体として,正答率はあまり変わりませんが,一致条件(congruent)の方が不一致条件(incongruent)よりも反応時間が短いことがわかりますね。私が適当に3回取り組んだ結果ですが,まあまあきれいな結果ですね。
db_df %>%
mutate(id = ifelse(ID == "2019_8_22_23_48_59_fxlmh9SL", "time1", ifelse(ID == "2019_8_22_23_56_32_juStKRbZ", "time2","time3"))) %>%
group_by(id,stim_type) %>%
summarise(正答率=sum(correct2,na.rm = TRUE)/length(correct2), 平均反応時間=mean(rt,na.rm = TRUE))## `summarise()` has grouped output by 'id'. You can override using the `.groups`
## argument.## # A tibble: 6 × 4
## id stim_type 正答率 平均反応時間
## <chr> <chr> <dbl> <dbl>
## 1 time1 congruent 1 781.
## 2 time1 incongruent 1 1028.
## 3 time2 congruent 1 769.
## 4 time2 incongruent 1 1113.
## 5 time3 congruent 1 888.
## 6 time3 incongruent 0.833 994.平均反応時間をプロットすると以下のような感じです。
db_df %>%
mutate(id = ifelse(ID == "2019_8_22_23_48_59_fxlmh9SL", "time1", ifelse(ID == "2019_8_22_23_56_32_juStKRbZ", "time2","time3"))) %>%
ggplot(aes(x=id,y=rt,fill=stim_type)) +
geom_bar(position = position_dodge(),stat = "identity") ## Warning: Removed 2 rows containing missing values (geom_bar).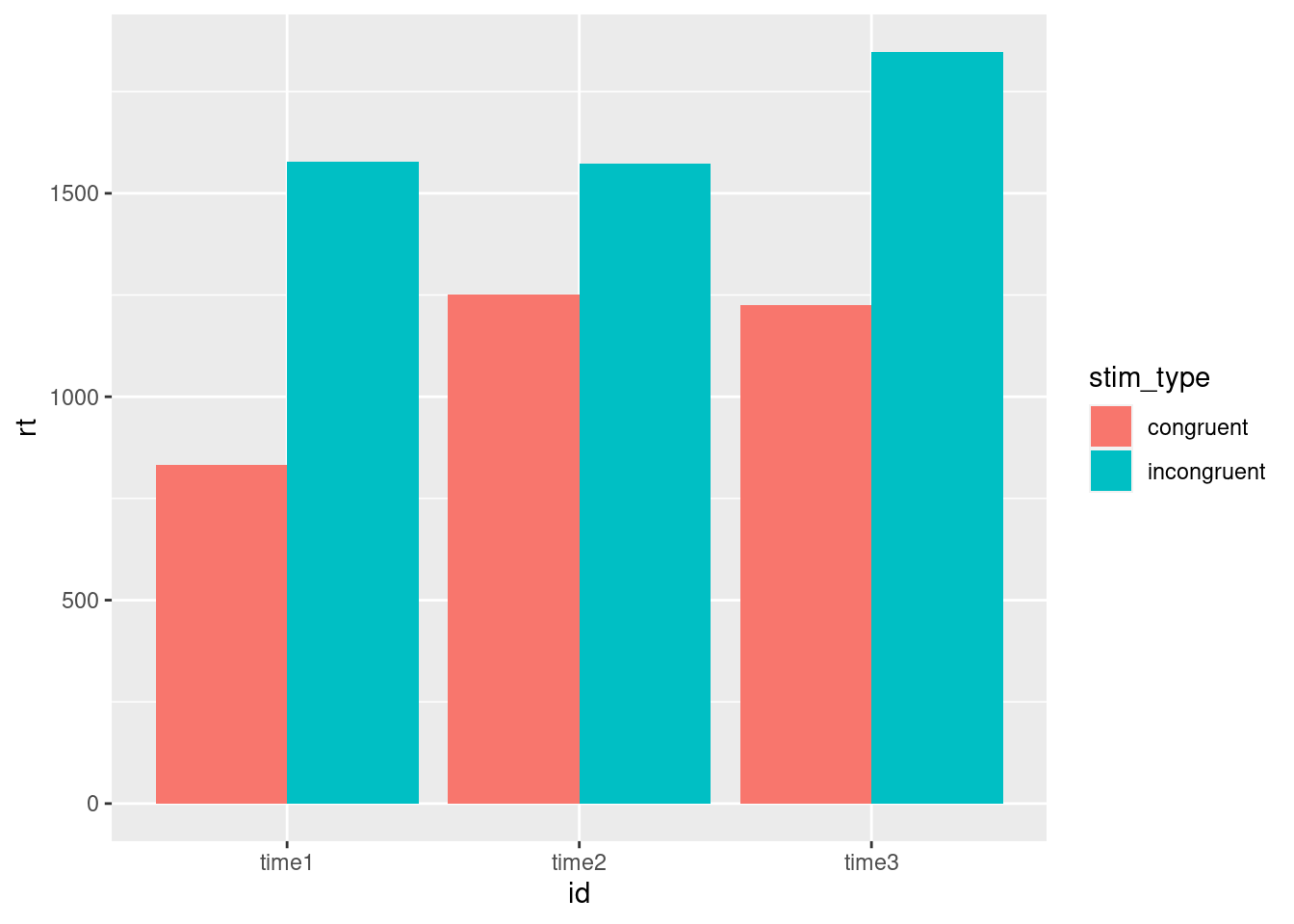
3 PsychoPyを用いた実験で得られたcsvファイルの処理
本研究室では,2020年度からjsPsychを使った認知課題の作成を学ぶようになっていますが,BIOPACやEEGを用いた実験などでは,PsychoPyで実験をしたほうが良いことがあります。PsychoPyでの実験では,参加者ごとにcsvファイルが得られます,以下では,そのようなcsvファイルの処理について解説しています。
4.1 データとフォルダの準備
PsychoPyを用いた実験のサンプルデータをクリックして,ダウンロードして,自分のパソコンの好きな場所に解凍・展開してください。“Analysis”フォルダ内に”Data”フォルダがあり,その中に,sub01.csvからsub04.csvという名前のcsvファイルが入っているかと思います。sub01.csvからsub04.csvは,逆転学習課題についての4名分の仮想データになります。RstudioのWorking Directoryにこの”Analysis”フォルダを置くか,“Analysis”フォルダのある場所をRstudioのWorking Directoryにしてください。
今回は,すでにフォルダに名前がついていましたが,自分でフォルダ名を付ける場合は,日本語を使用するのは推奨しません。フォルダとファイルと変数名には,英語(日本語のローマ字化でけっこう)を使用してください。
4.2 カレントディレクトリーの移動
基本的には,Analysisフォルダで解析の前処理や解析は行いますが,一時的にフォルダ移動する必要があります。その時に便利なのが,getwd()とsetwd()です。wdは,作業ディレクトリ(working directory)を意味し,getwdで現在の作業ディレクトリの情報を取得し,setwdで作業ディレクトリを設定します。
以下をRstudioのConsoleにタイプしてみましょう!
getwd()おそらく,さきほど設定したAnalysisフォルダまでのパスが出力されたかと思います(そうじゃない場合は,Rstudioのworking directoryを設定し直してください)。
次に,Analysisフォルダの1つ下の階層にあるDataフォルダに移動してみましょう。以下をRstudioのConsoleにタイプしてみましょう!
workDir <- getwd()
setwd(paste(workDir, "Data", sep = "/"))上記のコードでは,まず1行目で,getwd()で取得した作業ディレクトリのパスをworkDirに入れます。ここで,<-という矢印は,右側のものを左側のものにいれる(代入する)ということを意味します。次に,setwdを使って,workDir(作業ディレクトリのパス)とDataをpasteで結合し(sep=“/”で/で区切るように設定),Analysis下のDataフォルダに作業ディレクトリを変更しています。上記を打ち込んだら,getwd()をタイプして,ちゃんとDataフォルダが作業ディレクトリになっているか確認しましょう!
なお,DataフォルダからAnalysisフォルダに戻るには,以下のsetwd(“..”)が便利です(“..”で1つ上の階層に移動します)。
setwd("..")上記のsetwd(“..”)をRstudioのConsoleに打ちこんでから,getwd()でAnalysisフォルダに戻ってきているか確認をしてみましょう。
4.3 Dataフォルダ内のファイル名を取得
もう一度,Dataフォルダに移動します。その上で,list.filse()を使って,フォルダ内のファイル名をリスト化します(そしてfileNamesに入れる)。
workDir <- getwd()setwd(paste(workDir, "Data", sep = "/"))
fileNames <- list.files()Analysisフォルダに”..”で戻ります。
setwd("..")fileNamseの中身を確認します。フォルダに入っているファイル名がfileNamesに格納されているかと思います。
print(fileNames)## [1] "sub01.csv" "sub02.csv" "sub03.csv" "sub04.csv"4.4 Dataフォルダ内のファイル数を確認
fileNamesに格納されているファイルの数から参加者数を確認します。lengthでデータの長さ(ここでは,fileNamesに含まれるデータの個数)がわかります。今回は,4個のデータが入っています。
numberSubject <- length(fileNames)
print(numberSubject)## [1] 44.5 sub01のデータを読み込んでみる
では,早速,sub01.csvを読み込んでみましょう!tidyverseに入っているreadrパッケージのread_csv()でcsvファイルが読み込めます。そして,読み込むファイル名は,fileNamesに格納されているのものの1つ目(つまり,sub01)です。
sub01 <- read_csv(paste(workDir, "Data",fileNames[1], sep = "/"))
sub01## # A tibble: 122 × 8
## rt key_press trial_type trial_index time_elapsed internal_node_id correct
## <dbl> <dbl> <chr> <dbl> <dbl> <chr> <lgl>
## 1 8693 32 text 0 8697 0.0-0.0 NA
## 2 1194 90 categorize 1 12904 0.0-1.0-0.0 TRUE
## 3 316 90 categorize 2 16231 0.0-1.0-1.0 TRUE
## 4 313 90 categorize 3 19552 0.0-1.0-2.0 FALSE
## 5 352 77 categorize 4 22911 0.0-1.0-3.0 FALSE
## 6 450 90 categorize 5 26367 0.0-1.0-4.0 TRUE
## 7 298 90 categorize 6 29672 0.0-1.0-5.0 TRUE
## 8 305 90 categorize 7 32985 0.0-1.0-6.0 TRUE
## 9 197 90 categorize 8 36193 0.0-1.0-7.0 TRUE
## 10 296 90 categorize 9 39495 0.0-1.0-8.0 FALSE
## # … with 112 more rows, and 1 more variable: stimulus <chr>4.6 sub01のデータで必要な部分を抽出
上記のsub01のデータの解析で必要なのは,rt(反応時間), key_press(押したキー),trial_index(試行番号),correct(正誤)になります。また,trial_typeでは,categorizeのデータだけが欲しい(textは教示なので,いらない)。このようなデータの整理では,tidyverseのdplyrパッケージを使います。ここで,%>%というパイプ演算子というものがでてきます。これは,%>%の左(or前)のものが,次にくる関数の第一引数に入ることを意味します(例えば,上のlength(fileNames)の場合,length()関数の第一引数は,ファイル名リストのfileNamesになります)。
具体的な操作は以下になります。(1)まず,filterで,trial_typeが”categorize”なものにしぼります。(2)key_pressは,90だと紫の選択,77だと緑の選択になります。90と77だと扱いにくいので,90は1,77は0に変換します。ifelse(key_press == 90, 1,0)を使って,90なら1,それ以外は0になるようにして,mutateの新しい変数rewardを作成します(mutateは,新しい変数を作成する関数です)。(3)correctは,true,falseになるのですが,これも扱いにくいので,trueは1,falseは0にします。as.numeric(as.logical(correct))を使って,論理値にした上で,数値型に変換します。これもmutateを使って新たにrewardという名前を付けます。(4)この段階で,key_press, trial_type, time_elapsed, internal_node_id, correct, stimulusが要らなくなりました。selectを使って,不要な変数にマイナスをつけて除外します。(5)最後に,renameを使ってtrial_indexをnoに変更します。
# データの整理
sub01 <- sub01 %>%
filter(trial_type=="categorize") %>%
mutate(choice = ifelse(key_press == 90, 1,0)) %>%
mutate(reward = as.numeric(as.logical(correct))) %>%
select(-key_press, -trial_type, -time_elapsed, -internal_node_id, -correct, -stimulus) %>%
rename(no = trial_index)
# データの確認
sub01## # A tibble: 120 × 4
## rt no choice reward
## <dbl> <dbl> <dbl> <dbl>
## 1 1194 1 1 1
## 2 316 2 1 1
## 3 313 3 1 0
## 4 352 4 0 0
## 5 450 5 1 1
## 6 298 6 1 1
## 7 305 7 1 1
## 8 197 8 1 1
## 9 296 9 1 0
## 10 346 10 0 1
## # … with 110 more rowsこれで,必要な部分だけきれいに抽出できました。
4.7 4名のデータの読み込みと保存
4.7.1 データの読み込み
さて,今度は4名分のデータ(フォルダ内のすべてのデータ)を読み込んで,整理してみましょう。フォルダ内のデータ数分(今回は4)だけ,上記の操作を繰り返します。繰り返す場合は,for文を使います。for文は以下のように書きます。1から”繰り返す回数”までを順番にiに代入しつつ,“繰り返したい操作”を繰り返します。
for(i in 1:繰り返す回数){
繰り返したい操作
}csvファイルを繰り返し読み込むだけでは,それぞれをバラバラに読み込むだけになります。そこで,analysisDataという変数を作って,新たにデータを読み込んで・整理したら,rbind()を使って結合するとう作業をしてみましょう。
# analysisDataの準備
analysisData <- NULL
# 1からnumberSubject分(4回),操作を繰り返す
for(i in 1:numberSubject){
# 読み込んだcsvファイルのデータをtempDataに保存(tempは一時的を意味するtemporaryの省略です)。
tempData <- read_csv(paste(workDir, "Data",fileNames[i], sep = "/"))
# 上記とほぼ同じ操作をする(最後にデータ数分id番号を追加しています)
tempData <- tempData %>%
filter(trial_type=="categorize") %>%
mutate(choice = ifelse(key_press == 90, 1,0)) %>%
mutate(reward = as.numeric(as.logical(correct))) %>%
select(-key_press, -trial_type, -time_elapsed, -internal_node_id, -correct, -stimulus) %>%
rename(no = trial_index) %>%
mutate(id = rep(i,length(rt)))
#データの結合
analysisData = rbind(analysisData, tempData)
}
# データの確認
analysisData## # A tibble: 480 × 5
## rt no choice reward id
## <dbl> <dbl> <dbl> <dbl> <int>
## 1 1194 1 1 1 1
## 2 316 2 1 1 1
## 3 313 3 1 0 1
## 4 352 4 0 0 1
## 5 450 5 1 1 1
## 6 298 6 1 1 1
## 7 305 7 1 1 1
## 8 197 8 1 1 1
## 9 296 9 1 0 1
## 10 346 10 0 1 1
## # … with 470 more rows4.7.2 データの保存
これで,4名分の生データを読み込んだので,今度は,これをcsvファイルとして保存します。csvファイルは,write.csv()を使って,保存できます。
write.csv(analysisData, "analysisData.csv")4.7.3 IDの対応表
上記の作業では,for文でのiをIDとしているが,それが実際のどのファイルに対応するのか対応づけできてない。そこで,以下では,上記と同じようなfor文を使って,iとフォルダのファイル名との対応づけをした対応表(idTable)を作成する。また,tidyverseのstringrパッケージを用いると(stringrパッケージはtidyverseを読み込むだけでは読み込まれないので,別途libraryで読み込む),文字列の一部を抽出することもできる。今回は,参加者ID番号にかかわる4~5番目の文字を抽出してみて,それをidTableにいれる。
library(stringr)
idTable <- NULL
for(i in 1:numberSubject){
idTable$csvName[i] <- fileNames[i]
# csvのファイル名の4~5文字目を抽出(つまりsubと.csvの間のID番号の部分)
idTable$csvId[i] <- str_sub(fileNames[i],4,5)
idTable$dataId[i] <- i
}
idTable <- as.data.frame(idTable)
print(idTable)## csvName csvId dataId
## 1 sub01.csv 01 1
## 2 sub02.csv 02 2
## 3 sub03.csv 03 3
## 4 sub04.csv 04 44.8 もう少し処理をしたデータの読み込みと保存
4.8.1 データの読み込み
上記では,フォルダ内のすべてのcsvファイルを読み込んで,その生データを結合して,1つのcsvファイルで保存しました。私個人としては,このように各試行ごとのデータを使って解析した方が良いと考えますが,目的によっては,各試行ごとのデータではなく,全試行における正答率などを参加者ごとに計算した場合もあるかと思います。今回は,各参加者のデータを読み込んで,紫を選んだ比率(1が紫,緑は0)と正答率(correctでは1が正答,0が誤答)を各参加者ごとにsummaryDataに保存してみます。先程ののanalysiDataを作成したコードにちょっとだけ追加をしてみます。
# analysisDataの準備
analysisData <- NULL
# summaryDataの準備
summaryData <- NULL
# 1からnumberSubject分(4回),操作を繰り返す
for(i in 1:numberSubject){
# 読み込んだcsvファイルのデータをtempDataに保存(tempは一時的を意味するtemporaryの省略です)。
tempData <- read_csv(paste(workDir, "Data",fileNames[i], sep = "/"))
# 上記とほぼ同じ操作をする(最後にデータ数分id番号を追加しています)
tempData <- tempData %>%
filter(trial_type=="categorize") %>%
mutate(choice = ifelse(key_press == 90, 1,0)) %>%
mutate(reward = as.numeric(as.logical(correct))) %>%
select(-key_press, -trial_type, -time_elapsed, -internal_node_id, -correct, -stimulus) %>%
rename(no = trial_index) %>%
mutate(id = rep(i,length(rt)))
#データの結合
analysisData = rbind(analysisData, tempData)
# ここまで一緒。ここから,summaryDataの追加作業
summaryData$id[i] <- i
# 紫の選択率
summaryData$purpleRate[i] <- sum(tempData$reward)/length(tempData$reward)
# 正答率
summaryData$correctRate[i] <- sum(tempData$choice)/length(tempData$choice)
}
#データフレーム化と結果の表示
summaryData <- as_data_frame(summaryData)## Warning: `as_data_frame()` was deprecated in tibble 2.0.0.
## Please use `as_tibble()` instead.
## The signature and semantics have changed, see `?as_tibble`.
## This warning is displayed once every 8 hours.
## Call `lifecycle::last_lifecycle_warnings()` to see where this warning was generated.summaryData## # A tibble: 4 × 3
## id purpleRate correctRate
## <int> <dbl> <dbl>
## 1 1 0.633 0.583
## 2 2 0.483 0.55
## 3 3 0.483 0.567
## 4 4 0.575 0.6254.9 データの保存
summaryDataをcsv形式で保存する。
write.csv(summaryData, "summaryData.csv")